こんにちは、リブセンスでデータサイエンティストをしている北原です。今回も前回に引き続きアウトカムが2値のHeterogeneous Treatment Effects(HTE)に関する簡単な検証実験を扱います。ベイズを利用してT-Learnerに事前知識を組み込むことで推定が改善されるのかを調べたものです。コードはRとStanです。前回の記事は以下。
データ
データは前回の記事で利用したものを流用します。また、比較用に前回の記事でも扱ったロジスティック回帰ベースのT-Learnerを利用します。前回記事に掲載したものとほぼ同じですが、今回利用するコードを再掲します。
library(tidyverse) # Stan関係 library(cmdstanr) library(tidybayes) library(doParallel) registerDoParallel(detectCores()) options(mc.cores = parallel::detectCores()) set.seed(1) sig <- function(x) { 1 / (1 + exp(-x)) } make_data <- function( z_p, # 処置比率のパラメータ t_p, # 処置効果の大きさのパラメータ N = 1000) { # 共変量(1つ) x <- runif(n = N, min = -1, max = 1) # 処置 s_z <- z_p + 2 * x z <- rbernoulli(n = N, p = sig(s_z)) %>% as.integer() # 処置効果の大きさ tr <- t_p * x # 結果変数 s_y <- -1 + tr * z + 5 * x y <- rbernoulli(n = N, p = sig(s_y)) %>% as.integer() # HTE hte <- sig(-1 + tr + 5 * x) - sig(-1 + 5 * x) tibble(x, z, tr, y, hte) } # 処置比率のパラメータ z_ratio_parameter <- c(0, -1.1, -2.6) z_ratio_caption <- c("処置5割", "処置3割", "処置1割") # 処置効果の大きさのパラメータ tr_parameter <- c(9, 4, 1, -1, -2) # サンプルデータセット d <- tibble(id = 1, z_ratio_parameter, z_ratio_caption) %>% full_join(tibble(id = 1, tr_parameter), by = "id") %>% select(-1) %>% mutate(id = 1:n()) %>% select(id, z_ratio_parameter, z_ratio_caption, tr_parameter) %>% mutate(data = map2(z_ratio_parameter, tr_parameter, make_data)) # ロジスティック回帰ベースのT-Learnerの推定結果 t_learner_lr <- function(d) { m0 <- glm(y ~ x, d %>% filter(z == 0), family = "binomial") m1 <- glm(y ~ x, d %>% filter(z == 1), family = "binomial") predict(m1, d, type = "response") - predict(m0, d, type = "response") } d_lr <- d %>% mutate(est = map(data, t_learner_lr)) %>% mutate(data_est = map2(data, est, ~ mutate(.x, lr = .y))) %>% unnest(data_est)
T-Learnerのベイズ推定(事前知識なし)
まず、特に事前知識を入れずにStanでT-Learnerを実装します。StanコードとHTE推定までのRコードは以下の通りです。
// t-leaerner.stan
data {
int<lower=0> N0;
array[N0] int<lower=0,upper=1> y0;
vector[N0] x0;
int<lower=0> N1;
array[N1] int<lower=0,upper=1> y1;
vector[N1] x1;
vector[N0 + N1] x;
}
parameters {
real a0;
real b0;
real a1;
real b1;
}
model {
y0 ~ bernoulli_logit(a0 + b0 * x0);
y1 ~ bernoulli_logit(a1 + b1 * x1);
}
generated quantities {
vector[N0 + N1] hte;
for (i in 1:(N0 + N1)) {
hte[i] = bernoulli_logit_rng(a1 + b1 * x[i]) - bernoulli_logit_rng(a0 + b0 * x[i]);
}
}
t_learner_bayes <- function(d, stan_code_fname) { d0 <- d %>% filter(z == 0) d1 <- d %>% filter(z == 1) model_tlerner <- cmdstan_model(stan_code_fname) model_tlerner$sample( data = list( N0 = nrow(d0), y0 = d0$y, x0 = d0$x, N1 = nrow(d1), y1 = d1$y, x1 = d1$x, x = d$x ), seed = 1) } d_bayes <- d %>% mutate(fit = map(data, ~ t_learner_bayes(., "t-learner.stan"))) %>% mutate(fit_tidy = map(fit, ~ spread_draws(., a, b0, b1, hte[i])))
設定したHTEやロジスティック回帰ベースのT-Learnerの推定結果との比較は以下の通りです。
# 通常のロジスティック回帰ベースとの比較 d_bayes %>% mutate(est = map(fit_tidy, ~ group_by(., i) %>% summarise(hte = mean(hte)) %>% pull(hte))) %>% mutate(data_est = map2(data, est, ~ mutate(.x, bayes = .y))) %>% unnest(data_est) %>% select(z_ratio_caption, tr_parameter, x, hte, bayes) %>% bind_cols(d_lr %>% select(lr)) %>% rename(正解 = hte) %>% pivot_longer(cols = 正解:lr, names_to = "type", values_to = "推定結果") %>% ggplot(aes(x = x, y = 推定結果, group = type, color = type)) + geom_point() + facet_grid(z_ratio_caption ~ tr_parameter)
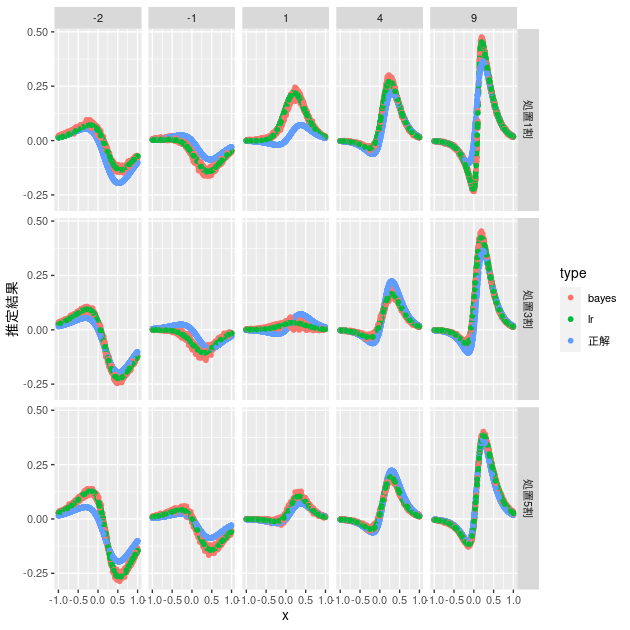
ベイズでも通常のロジスティック回帰ベースとほぼ同じ結果になっていることがわかります。説明変数が一つでオーバーフィットも生じにくいので、基本的なモデルが同じままベイズ推定を行なってもほぼ同じ結果になります。これぐらいのデータで推定結果を改善することが目的であれば、事前知識を導入しない場合はあえてベイズを利用する必要はなさそうです。
T-Learnerのベイズ推定(事前知識を導入)
ベイズを使う利点は、事前知識を容易に組み込むことができるところにあります。そこで、事前知識を少し加えて、前節のベイズモデルを改善してみましょう。
今回の問題設定では処置効果が属性に依存していることが前提です。したがって、属性が同じサンプルについてはHTEが0となるはずです。モデルとしては、処置ありとなしのロジスティック回帰モデルの定数項を同じものにすることで表現できます。この仮定を加えたStanコードは以下です。
// t-learner2.stan
data {
int<lower=0> N0;
array[N0] int<lower=0,upper=1> y0;
vector[N0] x0;
int<lower=0> N1;
array[N1] int<lower=0,upper=1> y1;
vector[N1] x1;
vector[N0 + N1] x;
}
parameters {
real a; // 定数項を共通化
real b0;
real b1;
}
model {
y0 ~ bernoulli_logit(a + b0 * x0);
y1 ~ bernoulli_logit(a + b1 * x1);
}
generated quantities {
vector[N0 + N1] hte;
for (i in 1:(N0 + N1)) {
hte[i] = bernoulli_logit_rng(a + b1 * x[i]) - bernoulli_logit_rng(a + b0 * x[i]);
}
}
前節と同じように、設定したHTEやロジスティック回帰ベースと比較した結果は以下のようになります。
d_bayes2 <- d %>% mutate(fit = map(data, ~ t_learner_bayes(., "t-learner2.stan"))) %>% mutate(fit_tidy = map(fit, ~ spread_draws(., a, b0, b1, hte[i]))) d_bayes2 %>% mutate(est = map(fit_tidy, ~ group_by(., i) %>% summarise(hte = mean(hte)) %>% pull(hte))) %>% mutate(data_est = map2(data, est, ~ mutate(.x, bayes = .y))) %>% unnest(data_est) %>% select(z_ratio_caption, tr_parameter, x, hte, bayes) %>% bind_cols(d_lr %>% select(lr)) %>% rename(正解 = hte) %>% pivot_longer(cols = 正解:lr, names_to = "type", values_to = "推定結果") %>% ggplot(aes(x = x, y = 推定結果, group = type, color = type)) + geom_point() + facet_grid(z_ratio_caption ~ tr_parameter)
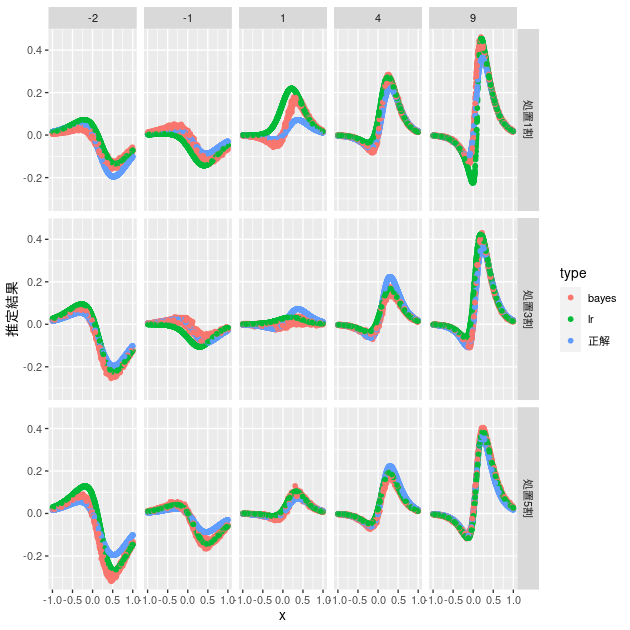
事前知識なしではロジスティック回帰ベースとほぼ同じ推定結果になっていましたが、定数項を共通化した結果、設定したHTEに近い推定結果が得られやすくなっていることがわかります。特に処置比率が5割から乖離するほど、処置効果が小さいほど、ロジスティック回帰ベースよりも設定したHTEに近い推定結果が得られやすくなっているようです。このようなケースではデータだけでは十分な推定が難しいため、事前知識が有効に機能するのかもしれません。
まとめ
今回はベイズ推定を利用したT-Learnerの検証を行いました。特に何も事前知識を入れないとベイズを利用する利点はあまりなかったですが、事前知識を適切にモデルに組み込むことができると推定結果を改善できることが確認できました。因果推論としては邪道な気がしますが、KPIを向上させることができるのならばこういう方法も実務上の利用価値はあると思います。
